4.5.10 分类项-对组排异损失(N-Pair Loss)
迭代公式:
图像:
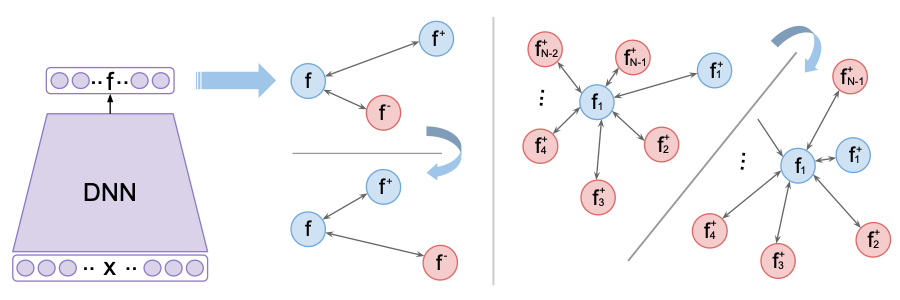
图 4-34 N-Pair Loss 函数图[16]
特性:
- 使具有相同标签的样本(positive)之间的距离,尽量接近
- 使具有不同标签的样本(negative)之间的距离,尽量远离
- 输入 N+1 个子集:1 个相似正样本集、N-1 个相反负样本集、1 个原样本对照集
- 同三元组损失一样,输入样本集,都在同权重下并行训练
- 以 项代表二维平面上多角度排斥最小力矩,一般 防止样本过近重合
- 越接近目标,损失越小
- 光滑(smooth),适合优化算法
- 非指数计算,算力消耗相对较低
对组排异损失(N-Pair Loss) 的提出,旨在解决 对比损失(Contrastive Loss) 和 三元组损失(Triplet Loss) 在分类上的局限性问题 [16] 。这两者,从物理学受力角度分析权重促成的样本聚集,会发现都是一维运动过程。
N-Pair Loss 的使用
N-Pair Loss 在每一次计算中,采用了 同样本集(Positive Set) 和 负样本类集(Negative Classes Set) 的概念。类集中的每一个负样本,都会对预测结果产生排斥,而单一的正样本则会对预测结果产生吸引。这样就能够更好地实现同类型聚集效果。一个比较适当的例子,就像一滴油散到了水中,最终会被水排斥而聚成一个集合的油滴。
实际上,基克·索恩(Kihyuk Sohn) 在对组排异损失的推导过程中,详细描述了从 Triplet Loss -> (N+1)-Tuplet Loss -> N-Pair Loss 的完整过程。其中,(N+1)-Tuplet Loss 可认为是 N-Pair Loss 的过渡。
文中指出,当 N = 2 时,(N+1)-Tuplet Loss 可认为近似于 Triplet Loss。以此为起点,我们很快便会发现,对组排异损失 相当于将 三元组损失中 一组 相似正样本集(Positives) 、 一组 相反负样本集(Negatives) 、 一组 原样本对照集(Anchors) 总共三组之间,两两样本集间样本的距离均值计算,改换成了 一组 相似正样本集(Positives) 、 多组 相反负样本集(Negatives) 、 一组 原样本对照集(Anchors) 总共 N+1 组之间的距离计算。
即,相较于三元组损失,进一步细化了 相反负样本集(Negatives)内,不同标签的对正样本集的驱动作用。
同样以人脸识别(FD [Face Detection])为例,由 位不同人的 张该人不同脸的图片样本组成的,样本总量 大小的数据集。
对组排异损失要求,也是三种样本分类子集分类:
- 相似正样本集(Positives),由同人不同脸组成的 大小子集
- 相反负样本集(Negatives),由不同人不同脸组成的 组各 大小子集
- 原样本对照集(Anchors),由不同人同脸(选一校订)组成的 大小子集
这三类子集,在数据分批后,会被分为相同批数并组合为一批数据,作为单次迭代输入数据,参与训练。我们仍然采用角标 来表示分批,那么有:
则,在分批数据参与一次批计算后,最终还是会构成同三元组损失类似的 大小的一组嵌入集(Embeddings),被我们用来计算损失函数(Loss)的实际处理对象。
因此,在工程上,我们 只需要更换单次损失的计算公式,就能从三元组损失的迁移至对组排异损失的计算过程。
N-Pair Loss 算子化
利用 C 语言实现对算子的封装,有:
#include <math.h>
#include <stdbool.h>
#include <stdio.h>
#include <stdlib.h>
#define BATCH_SIZE 10 // Batch_size = Samples_of_Person x Data/Person
#define VECTOR_SIZE 128 // Extract output layer Feature vector's dimissions
#define DEVIDE_SAFE 1e-12 // protect when gridant at 0 will be to lage
// Pairwise Distance Calculation
void pairwise_distance(double embeddings[BATCH_SIZE][VECTOR_SIZE],
double distances[BATCH_SIZE][BATCH_SIZE], bool squared) {
for (int i = 0; i < BATCH_SIZE; i++) {
for (int j = 0; j < BATCH_SIZE; j++) {
double dot_product = 0.0;
double square_norm_i = 0.0;
double square_norm_j = 0.0;
for (int k = 0; k < VECTOR_SIZE; k++) {
dot_product += embeddings[i][k] * embeddings[j][k];
square_norm_i += embeddings[i][k] * embeddings[i][k];
square_norm_j += embeddings[j][k] * embeddings[j][k];
}
distances[i][j] = square_norm_i - (2.0 * dot_product) + square_norm_j;
if (!squared) {
if (distances[i][j] < 0.0) {
distances[i][j] = 0.0;
} else {
distances[i][j] = sqrt(distances[i][j]);
}
}
}
}
}
// Get N-Pair Mask (Totally same with Triplet Mask)
void get_n_pair_mask(int labels[BATCH_SIZE],
bool mask[BATCH_SIZE][BATCH_SIZE][BATCH_SIZE]) {
for (int i = 0; i < BATCH_SIZE; i++) {
for (int j = 0; j < BATCH_SIZE; j++) {
for (int k = 0; k < BATCH_SIZE; k++) {
// indices_equal
bool i_not_j = (i != j);
bool i_not_k = (i != k);
bool j_not_k = (j != k);
bool distinct_indices = (i_not_j && i_not_k && j_not_k);
// label_equal
bool i_equal_j = (labels[i] == labels[j]);
bool i_equal_k = (labels[i] == labels[k]);
bool valid_labels = (i_equal_j && !i_equal_k);
// mask depends on both
mask[i][j][k] = (distinct_indices && valid_labels);
}
}
}
}
// Batch All N-Pair Loss
double n_pair_loss(int labels[BATCH_SIZE],
double embeddings[BATCH_SIZE][VECTOR_SIZE], double margin,
bool squared, double *fraction_positives) {
// So, this only caused once per epoch for certain storage space
double pairwise_distances[BATCH_SIZE][BATCH_SIZE];
bool n_paired_avail_masks[BATCH_SIZE][BATCH_SIZE][BATCH_SIZE];
// Pairwise distance calculation
pairwise_distance(embeddings, pairwise_distances, squared);
// Get n_pair mask
get_n_pair_mask(labels, n_paired_avail_masks);
// n_pair loss calculation
int num_positive = 0;
int num_validate = 0;
double n_pair_cost = 0.0;
{
for (int i = 0; i < BATCH_SIZE; i++) { // i for Anchor
double n_pair_loss = 0.0;
for (int j = 0; j < BATCH_SIZE; j++) { // j for positive
for (int k = 0; k < BATCH_SIZE; k++) { // k for negative
double current_mask = n_paired_avail_masks[i][j][k];
double current_loss = current_mask * exp(pairwise_distances[i][j] -
pairwise_distances[i][k]);
n_pair_loss += current_loss;
// Calculate number of positive n_pairs and valid n_pairs
if (current_loss > margin) {
num_positive++;
}
if (current_mask > 0) {
num_validate++;
}
}
}
n_pair_cost += log(margin + n_pair_loss);
}
}
// Calculate fraction of positive n_pairs
*fraction_positives =
(double)num_positive / ((double)num_validate + DEVIDE_SAFE);
return n_pair_cost / (double)(num_positive + DEVIDE_SAFE);
}
int main() {
// Example input (fulfill to BATCH_SIZE x VECTOR_SIZE)
// Use Random labels and embeddings for testing
// Use three classes as different type, to generate labels
int type = 3;
int labels[BATCH_SIZE];
double embeddings[BATCH_SIZE][VECTOR_SIZE];
for (int i = 0; i < BATCH_SIZE; i++) {
labels[i] = rand() % type;
for (int j = 0; j < VECTOR_SIZE; j++) {
embeddings[i][j] = (double)rand() / (double)RAND_MAX;
}
}
double margin = 1.0;
double fraction_positives = 0.0;
double n_pair_cost_value =
n_pair_loss(labels, embeddings, margin, false, &fraction_positives);
printf("The n_pair loss is %f with positives %f \n", n_pair_cost_value,
fraction_positives);
return 0;
}
运行验证可得到结果:
The n_pair loss is 0.408567 with positives 0.377907
对组排异损失从样本宏观角度,统一了正负样本概念。指明了,非当前指向类的负样本,可以被认为是指向负样本类型情况的正样本。因此,对于 N 分类处理过程,整个运算损失计算时间复杂度被化简为仅有 2N。相当的高效。